COMO INTERPRETAR
O RELATÓRIO
GENÉTICO DE
SEU FILHO
Ajuda para familiares da Síndrome de Phelan-McDermid
CONTEÚDO DESTA PÁGINA
• Parte 1 – Nocões básicas sobre termos que aparecem em relatórios genéticos
A) Cromossomos, genes e as proteínas que eles produzem
B) Tipos de testes genéticos
C) Variantes e mutações
D) Fenótipo e penetrância
• Parte 2 – Como entender a linguagem técnica para uma variante de sequência
• Parte 3 – Como entender a linguagem técnica para uma exclusão (deleção)
• Parte 4: Recursos adicionais
Esta é uma tradução livre e autorizada de um guia elaborado pela Dra. Tesi Kohlenberg e pelo neurocientista Andrew Mitz. O guia original pode ser acessado no link abaixo:
“Esta visão geral tem como objetivo tornar os relatórios genéticos mais fáceis de ler para os pais. Ele foi escrito com os pais de crianças com PMS em mente, mas pode ser útil para outras pessoas. Não é um substituto para a consulta com geneticistas ou conselheiros genéticos; em vez disso, foi escrito para ajudar a tornar a conversa com profissionais médicos mais fácil e produtiva. ” — Teresa M. Kohlenberg, MD
PARTE 1 : Noções básicas sobre termos que aparecem em relatórios genéticos
A) Cromossomos, genes e as proteínas que eles produzem

• Cromossomo
Um cromossomo é uma longa cadeia de DNA enrolada. O DNA fornece instruções para todas as partes e funções de nossos corpos. Os humanos têm 46 cromossomos que vêm em pares (chamados de “autossomos” e têm números de 1 a 22) mais dois cromossomos sexuais que não estão em pares, X e Y. Os homens têm um X e um Y. As mulheres têm dois cromossomos X.
• Ácidos nucleicos (nucleotídeos)
Os nucleotídeos são os blocos de construção do DNA. Eles são arrajnados em pares, formando uma longa cadeia retorcida (chamada de dupla hélice). Existem apenas quatro nucleotídeos no código do DNA humano, geralmente escritos pelas primeiras letras de seus nomes: A = adenina, C = citosina, G = guanina, T = timina.
• Gene
Um gene é uma sequência de ácidos nucléicos (nucleotídeos) ao longo do DNA. A sequência é um código de como fazer proteínas. As proteínas são construídas a partir de aminoácidos. Cada grupo de 3 nucleotídeos sinaliza um aminoácido específico, por isso é crucial que uma sequência de gene seja transcrita e usada em grupos de 3.
• Aminoácidos
Os aminoácidos são os blocos de construção de proteínas que se ligam com base no código de DNA de um gene. As proteínas são o tijolo e a argamassa do corpo: elas formam as células e os órgãos do corpo. Proteínas especiais (enzimas) gerenciam as reações químicas do corpo. A maioria dos neurotransmissores e hormônios são proteínas. Existem cerca de 20 aminoácidos usados para fazer proteínas, cada um tipicamente identificado por uma abreviatura de 3 letras.
• RNA
Esta visão geral enfoca o código do DNA e as proteínas resultantes. No entanto, existem várias etapas intermediárias. O DNA está em uma zona central especial da célula chamada “núcleo”. O mecanismo de produção de proteínas está fora do núcleo, em uma zona chamada “citosol”. Para copiar as instruções do DNA, movê-las para fora do núcleo e, em seguida, usar as instruções para fazer muitas cópias da proteína, a célula usa o RNA. O processo é complexo e envolve vários tipos diferentes de RNA (tRNA, mRNA, rRNA). Esta visão geral não inclui as etapas do RNA.
B) Tipos de testes genéticos
• Cariótipo
A cariotipagem envolve abrir uma célula e espalhar os próprios cromossomos em uma lâmina para estudar ao microscópio. O observador pode contar o número de cromossomos e notar qualquer coisa incomum em sua forma. Por exemplo, o cariótipo é o método preferido para detectar um cromossomo em anel ou um cromossomo extra, como a cópia extra do cromossomo 21, que causa a Síndrome de Down, ou um cromossomo ausente, como na Síndrome de Turner. Embora os microarrays cromossômicos (veja abaixo) possam detectar pequenas mudanças não visíveis com o cariótipo, o cariótipo pode demonstrar a estrutura de deleções, duplicações, inserções, translocações desequilibradas, cromossomos em anel e outras anormalidades que um microarray pode não perceber.
• Hibridização in situ
Às vezes, “ish” ou FISH aparece em um relatório genético. “.Ish” significa “hibridização in situ”. É um método mais antigo para estudar exclusões e outros rearranjos. Às vezes, ainda é usado como um teste definitivo para certos rearranjos (por exemplo, translocação balanceada)..
• Microarray cromossômico
Frequentemente abreviado como “CMA”, é um teste sensível que procura segmentos cromossômicos extras (duplicados) ou ausentes (excluídos).
• Sequenciamento
O sequenciamento é uma técnica que lê as letras do código do DNA em ordem, letra por letra. Isso fornece informações muito detalhadas sobre um ou vários genes.
• Sequenciamento completo do exoma (WES)
Sequenciamento dos segmentos mais relevantes de cada gene do DNA. Fornece uma visão ampla, mas detalhada de todos os genes em todos os cromossomos.
• Sequenciamento do genoma completo (WGS)
de todos os genes do DNA. É mais detalhado do que o WES.
C) Variantes e mutações

• Variante
Todos os humanos têm o mesmo conjunto de genes (cerca de 20.000 genes diferentes) em seu DNA, mas pessoas diferentes podem ter versões diferentes de qualquer gene. As diferentes versões de um gene são chamadas de “variantes”. As variantes são responsáveis por muitas diferenças em coisas como a cor do cabelo, se uma pessoa tem sardas ou a probabilidade de ela ser alta ou baixa.
• Variante comum
Variante comumente encontrada na população em geral. Variantes comuns tendem a ser benignas (não prejudiciais).
• Variante patogênica
Variante de um gene conhecido por estar associado a doenças.
• Variante benigna
Variante de um gene que não causou doenças no passado.
• Variante de significância desconhecida (VUS)
Termo usado quando você não tem informações sobre uma variante ou quando suspeita que a variante está causando um problema, mas você não tem evidências suficientes para classificá-la como “patogênica”.
• Variante de novo
A variante não foi encontrada nos pais, mas existe na criança.
• Mutação
Um termo às vezes usado para indicar uma mudança na sequência do DNA que pode causar danos ou distúrbios. Esse uso do termo foi recentemente substituído pelo termo “variante patogênica”.
• Mosaico, mosaicismo
Algumas das células testadas contém uma variante, outras células contém uma variante diferente.
• Variante da linha germinativa
Variante encontrada apenas nos espermatozoides ou óvulos dos pais, mas que não está presente nas outras células do corpo (somáticas) dos pais. Mutações em linhas germinativas (espermatozoides ou óvulos) explicam como, em raras ocasiões, os pais com teste negativo para uma variante patogênica podem ter vários filhos com uma variante patogênica.
• Variante do número da cópia / variação do número da cópia (CNV)
Segmentos cromossômicos duplicados ou ausentes às vezes são chamados de variantes do número de cópia (CNVs). Qualquer adição ou subtração ao código do DNA pode ser chamada de CNV. Não há tamanho mínimo ou máximo ao falar sobre CNVs, embora uma vez que um cromossomo inteiro seja duplicado ou excluído, a terminologia mude para variação do número de cromossomos.
D) Fenótipo e penetrância

• Fenótipo
A forma como o indivíduo aparenta e funciona.
• Penetrância
A frequência com que uma variante (ou outra anormalidade genética) realmente causa uma mudança no fenótipo, como uma doença. Uma variante altamente penetrante quase sempre leva à doença / distúrbio.
• Penetrância incompleta
Uma variante (ou outra anormalidade genética) que causa doença apenas às vezes. Essa é outra explicação de como, em raras ocasiões, os pais podem ter a variante, mas não ter o transtorno, enquanto seus filhos têm o transtorno.
• Dominante autossômico
Os humanos têm duas cópias de cada gene, exceto os genes dos cromossomos sexuais, que não são pareados. Dominante autossômico significa que uma variante patogênica é suficiente para causar a doença. As variantes patogênicas de SHANK3 são geralmente autossômicas dominantes. (SHANK3 é um gene importante na Síndrome de Phelan-McDermid porque uma variante pode ser altamente penetrante, patogênica e autossômica dominante.)
PARTE 2 : Como entender a linguagem técnica para uma variante de sequência

Vamos começar com este exemplo, uma pequena parte do gene SHANK3. A linha superior mostra os nucleotídeos, listados em grupos de três (que é como a célula os “lê”). A linha inferior mostra os aminoácidos que são montados em uma proteína com base na “receita” do DNA.

A construção da proteína está completa quando a sequência de DNA atinge um códon de parada (STOP códon). Na verdade, existem três códons de parada diferentes: TGA, TAA e TAG. Qualquer um deles encerrará a montagem da proteína. Há também um códon de início indicando onde o gene começa: ATG.
O relatório genético pode ter uma seção que começa com “c.”. O “c” localiza a mudança no código do DNA (nucleotídeos). O relatório também pode ter uma parte que começa com “p.”, que localiza a alteração na proteína (aminoácidos) causada pela alteração no DNA.
Por exemplo:
O nucleotídeo G foi alterado para o nucleotídeo A na localização 2594. Esta substituição pode ter 3 efeitos possíveis dependendo da sequência em 2594.
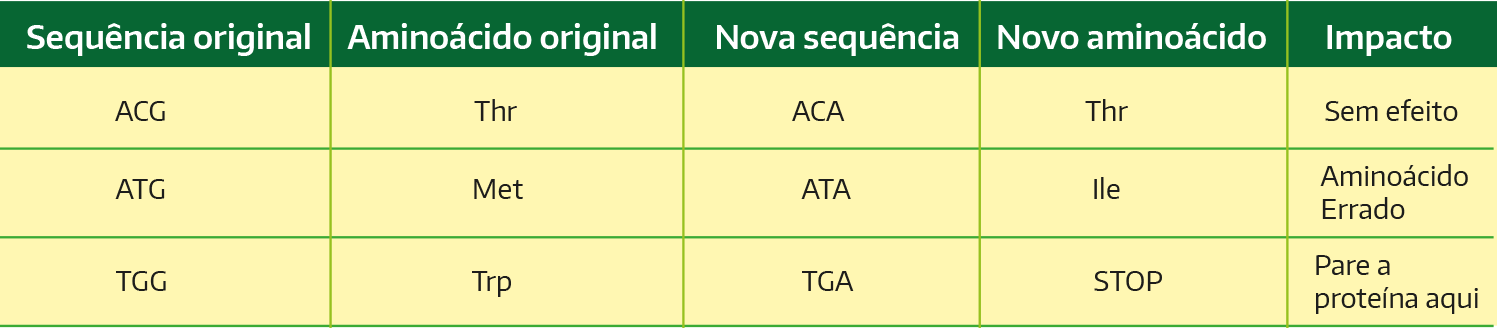
Os impactos dessas três possibilidades são provavelmente diferentes. No primeiro caso, geralmente não há impacto. No segundo caso, a mudança no aminoácido pode não ser importante ou pode desempenhar um papel crítico. No último caso, o código STOP truncará a proteína, o que geralmente a torna inutilizável. Todos os três casos são chamados de SNPs (pronuncia-se: “recortes”, polimorfismos de nucleotídeo único).
Variante: c.2594_2597dup
A barra de sublinhado aqui significa que a alteração afeta o intervalo dessas posições no código. “Dup” significa que as letras nas posições entre os dois números foram duplicadas. “Del” significa que eles foram excluídos.
Sempre que há uma mudança que não tem três letras (ou um múltiplo de três), há outro problema importante: a mudança muda o agrupamento de letras na leitura do código, o que distorce o código. Isso é chamado de frameshift. As mudanças de quadro geralmente causam grandes interrupções do gene.
No exemplo de um segmento do gene SHANK3 acima, uma deleção de dois nucleotídeos se parece com isto, se as duas letras que faltam forem do segundo grupo de três (destacado em vermelho):
Original:

Mudado:
Neste exemplo, dois nucleotídeos (C e T) estão faltando. O agrupamento de todas as letras é alterado automaticamente quando a sequência do gene é transcrita. A linha inferior mostra a nova sequência de aminoácidos:

Quando a proteína é produzida seguindo o código alterado, ela tem dois problemas: ela tem os aminoácidos errados e a TGA causará uma “PARADA” para sinalizar a terminação da proteína antes que ela deveria ter terminado.
p.Leu864Ala
Isso nos diz que o aminoácido Leu (Leucina) da sequência original foi alterado para um aminoácido Ala (Alanina). Em alguns casos, essa seria a única mudança. Em um frameshift, ele marca a primeira de uma série de alterações.
fs* 2
FS significa frameshift.
Um frameshift ocorre quando os grupos de 3 nucleotídeos foram deslocados ganhando ou perdendo 1, 2, 4 ou qualquer número não divisível por 3, como em nosso exemplo. O *22 mostra quantos nucleotídeos a mais são transcritos até que seja executado um código STOP. Assim que atinge o STOP, o resto do gene é ignorado.
Heterozigoto: As duas cópias do SHANK3 são diferentes. Isso pode ocorrer porque a cópia de SHANK3 herdada da mãe é diferente da cópia herdada do pai ou porque uma cópia é típica e a outra é uma variante incomum.
Homozigoto: Ambas as cópias do SHANK3 são iguais. Normalmente, isso significa que ambas as cópias são típicas, porque os humanos não podem sobreviver se ambas as cópias forem patogênicas.
PARTE 3 : Como entender a linguagem técnica para uma exclusão (deleção)
Em PMS, a maioria – mas não todas – as deleções cromossômicas removem parte ou todo o SHANK3. Acredita-se que o SHANK3 seja muito importante, mas uma deleção cromossômica em PMS costuma ser grande o suficiente para remover de 45 a 50 genes. Doze desses genes foram identificados como provavelmente importantes para o fenótipo de PMS. Relatórios genéticos para deleções cromossômicas usam termos diferentes dos descritos acima para variantes.
Genoma de referência: Uma vez que o DNA humano foi decodificado (“sequenciado”) para muitas pessoas diferentes, os cientistas montaram um genoma mestre (tabela da sequência normal de nosso código de DNA), chamado de genoma de referência. Com o passar dos anos, a referência foi sendo refinada. Muitos relatórios atuais são baseados no GRCh37/hg19. (Hg19 é uma referência do genoma humano #19). A “construção” mais recente do genoma humano é GRCh38/hg38.
Localização da variante do número da cópia: A localização da variação do número de cópias pode ser descrita de diferentes maneiras.
Exemplo 1: deleção terminal
Arr [GRCh37] 22q13.31q13.33 (44802524-51304566) x1
Arr: Indica que os resultados foram obtidos por teste de matriz.
22q13.31q13.33: Isso deve parecer um pouco familiar. 22q13 significa cromossomo 22, braço longo “q”, região 1, banda 3. As bandas são regiões claras e escuras alternadas ao longo do cromossomo que são distinguíveis dos segmentos adjacentes. As bandas têm sub-bandas. Nesse caso, o relatório diz que o arranjo afeta 22q13.31 a 22q13.33. (sub-bandas 31 a 33).
(44802524-51304566): Esta é a localização precisa no cromossomo 22. Os números fazem sentido apenas para um genoma de referência específico. Nesse caso, os números vêm de hg19.
x1: Isso diz que o teste de matriz de cromossomos encontrou a sequência para apenas um cromossomo naquela região. É uma boa indicação de que ocorreu uma deleção naquela região do outro cromossomo.
Tamanho da deleção: Como o elemento (44802524-51304566) fornece localizações exatas, podemos subtrair os dois números para calcular o tamanho da exclusão. Neste exemplo, a exclusão é de 6.502.042 pares de bases (6,5 Mb). (Como os nucleotídeos estão em pares, a contagem de pares de bases é igual à contagem de nucleotídeos.)
Deleção terminal: Quando a parte excluída do cromossomo se estende até o final do cromossomo (o “término”), a deleção é chamada de deleção terminal.
Deleção intersticial: Quando a exclusão não se estende até o final do cromossomo.
Genes OMIM: Genes implicados em vários distúrbios foram catalogados online. OMIM é o site “Online Mendelian Inheritance in Man”. É um ótimo recurso para ler sobre genes e distúrbios associados a genes. https://www.omim.org/
Herança: Ao pesquisar genes, é importante prestar atenção à “herança”. AR significa autossômico recessivo, o que geralmente significa que, mesmo se uma pessoa tiver uma variante anormal, ter uma variante “normal” (comum) é suficiente para prevenir o transtorno. AD significa autossômico dominante, o que geralmente significa que ter uma variante anormal é suficiente para causar o transtorno, mesmo se a pessoa também tiver uma variante normal. A síndrome de Phelan McDermid é uma doença autossômica dominante.
Existem outros distúrbios graves relacionados a genes na mesma região do cromossomo 22, que podem ocorrer em pessoas com PMS apenas se, após a exclusão de uma cópia de um gene, a cópia restante for uma variante patogênica. É raro que pessoas com PMS também tenham uma variante patogênica na segunda cópia de um gene.
46, XY
Este é um cariótipo normal para um homem obtido por análise cromossômica.
Exemplo 2: cromossomo em anel
46, XY, r (22) (p13q13)
r (22) (p13q13): r (22) significa que o cromossomo 22 formou um anel, o que é anormal. Um cromossomo em anel se forma a partir da exclusão das duas extremidades terminais do cromossomo, seguida pela fusão das extremidades quebradas. No cromossomo 22, p13 está na extremidade do braço curto e q13 está na extremidade do braço longo. É comum que falte parte do cromossomo em um anel em uma ou nas duas extremidades. Se a extremidade q13 não tiver parte do cromossomo, isso seria uma deleção 22q13.3.
Exemplo 3: translocação
46, XX, t (19; 22) (q13,42; q13,31)
t (19; 22) (q13,42; q13,31)
Esta é uma translocação entre o braço longo (q) do cromossomo 19 e o braço longo (q) do cromossomo 22. É uma translocação equilibrada. Nenhuma deleção (perda de material genético) é indicada. Um pai com este arranjo pode ser um “portador” de PMS. A prole pode (~ 25% de chance) herdar uma translocação desequilibrada. (veja o próximo exemplo)
der (22) t (22; 19) (q13,31; q13,42)
Esta translocação desequilibrada cria um cromossomo 22 derivado (anormal) feito da maior parte do cromossomo 22 e parte do cromossomo 19. Isso representa a perda de parte do cromossomo 22 (deleção do cromossomo 22), além de uma cópia extra de parte do cromossomo 19 (trissomia). É uma forma de PMS.
Parte 4: Recursos adicionais
Citogenética: Nomenclatura e Doença
https://www.cibmtr.org/Meetings/Materials/CRPDMC/Documents/2008/Nov2008/NavarroW_Cytogenetic.pdf
Aqui está um site com muito mais detalhes.
https://www.hgvs.org/mutnomen/standards.html
O navegador do genoma humano localiza genes e muitas outras características de cada cromossomo. Aqui está o navegador configurado para visualizar uma exclusão de terminal de 1,1 Mb do cromossomo 22.
Agradecimentos
Este guia não teria sido possível sem a experiência e a ajuda paciente de Andy Mitz e a generosidade de Katy Phelan, que analisou e comentou um rascunho anterior. Também é informado pelo trabalho árduo e cuidado criativo dos pesquisadores e médicos dedicados que se concentram nos desafios que nossas famílias enfrentam, e pela defesa, educação e inovação impulsionadas pela Fundação PMS.
Nossa rede de famílias e amigos te espera de braços abertos. Participe da AFSPM
Entre em contato e faça parte da nossa Associação :
e-mail: contato@pmsbrasil.org.br

E-mail:
contato@pmsbrasil.org.br
© 2020-2023 - Associação da Síndrome de Phelan-McDermid do Brasil




